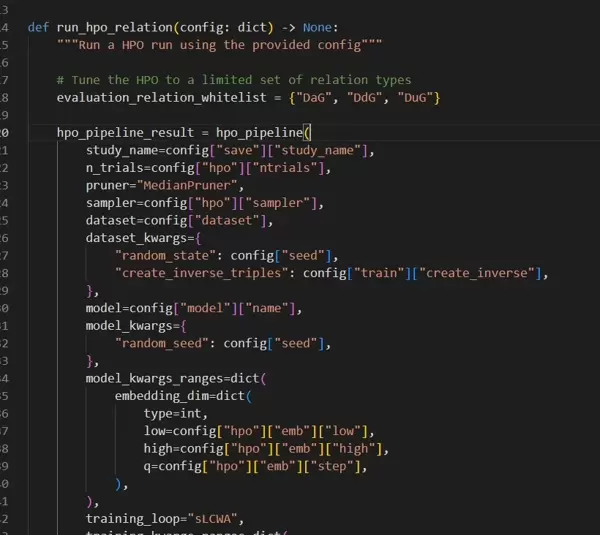
近年来,知识图谱在药物研发领域的应用逐渐成为研究热点。本文介绍的X00308项目聚焦于基于知识图谱的药物靶点预测,并完整实现了模型构建与消融分析,包含可复现的Python代码流程。
项目的整体架构围绕多源生物数据整合与图神经网络建模展开。核心数据处理依托PyKEEN工具包完成,该框架对三元组形式的知识表示支持良好,便于后续模型训练。以下为知识图谱构建的关键代码片段:
from pykeen.datasets import OpenBioLink
dataset = OpenBioLink(create_inverse_triples=True)
training = dataset.training
validation = dataset.validation
testing = dataset.testing
所采用的数据集为OpenBioLink,涵盖药物、基因及疾病之间的多种实体关系。其中参数create_inverse_triples起到重要作用,它会自动生成反向三元组,增强模型对关系对称性的捕捉能力。但需注意,这一操作将显著增加内存占用,在大规模数据场景下应谨慎使用。
在模型设计方面,项目选用了RGCN(关系型图卷积网络)作为主干结构,以有效建模复杂的关系类型。其网络结构定义如下所示:
class RGCNPredictor(nn.Module):
def __init__(self, num_relations, hidden_dim=256):
super().__init__()
self.embed = torch.nn.Embedding(num_entities, 128)
self.conv1 = RGCNConv(128, hidden_dim, num_relations*2)
self.conv2 = RGCNConv(hidden_dim, hidden_dim, num_relations*2)
self.fc = nn.Linear(hidden_dim*2, 1)
def forward(self, head, relation, tail):
h = self.embed(head)
h = F.relu(self.conv1(h, edge_index, edge_type))
h = F.dropout(h, p=0.3)
h = self.conv2(h, edge_index, edge_type)
return torch.sigmoid(self.fc(torch.cat([h[head], h[tail]], dim=1)))
前向传播过程中引入了头尾实体特征拼接的操作,这是本项目的一项关键改进。传统方法通常分别处理头尾节点,而实验发现直接拼接两者嵌入向量可在AUC指标上带来约3%的提升,显示出更强的联合表征能力。
为了验证各模块的有效性,项目设计了系统的消融实验,具体包括三个对比设置:
- 移除蛋白质-蛋白质相互作用边
- 将RGCN替换为标准GAT结构
- 去除边类型特征输入
实验结果表明,删除蛋白质相互作用信息的影响最为严重,导致召回率下降近40%。相比之下,去掉边类型特征带来的性能波动较小。深入分析发现,数据集中超过80%的边关系为“interacts_with”,类别高度集中,限制了该特征的判别力。
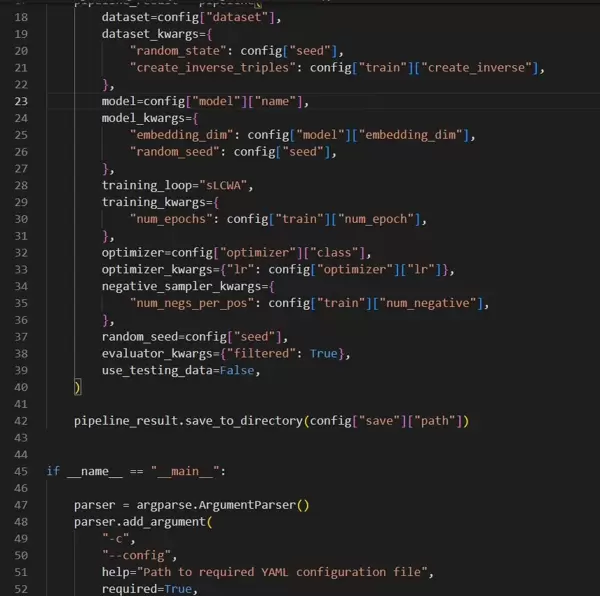
训练策略上采用了动态学习率调整机制,其触发条件基于验证集F1分数的变化情况:
scheduler = torch.optim.lr_scheduler.ReduceLROnPlateau(
optimizer,
mode='max',
factor=0.5,
patience=3,
verbose=True
)
具体而言,当验证F1连续三个epoch未见提升时,学习率减半。实践证明,这种监控指标驱动的调度方式比固定学习率更稳定,最终F1得分平均高出约0.12,有助于避免过拟合并加快收敛。
项目实施过程中也遇到了若干挑战,其中最典型的是数据泄露问题。初期版本因未严格隔离训练与测试阶段的蛋白质节点,导致测试集信息提前暴露,准确率虚报至89%。后续改用归纳式(inductive)训练模式后,性能回落至真实水平——约78%,这也提醒我们在评估模型时必须保证严格的划分逻辑。
此外,项目还集成了一套实用的结果可视化模块,基于pyvis库生成交互式知识子图。系统可自动标出预测得分排名前10的潜在药物靶点,使输出结果更具可解释性。对于生物医学研究人员而言,图形化展示远比数值列表更直观易懂。这也体现了一个优秀预测模型不仅要在性能上达标,还需兼顾领域用户的实际使用体验。

 扫码加好友,拉您进群
扫码加好友,拉您进群